어느 논문이나 거의 빠지지 않는 RNA-seq과 분석에 대해서 개인적으로 아주 간략, 빈약하게 적어보고자 한다.
(요즘 글이라는 것 자체를 쓰지를 않기 때문에 정보 전달보다는 어느 정도의 memory restoration/rehabilitation의 개념으로서 ㅎㅎ)
+) 나는 bioinformatics에 대한 지식은 없고 그저 user일뿐이다 (used to be an user)
먼저, 목적은 각 실험 또는 연구 내용에 따라서 달라질 것이지만,
보통 illumina의 Hiseq platform으로 whole RNA seq을 한다.
예컨대, sample을 준비하고 illumina 회사에 맡기거나 각 연구소/학교의 core lab에 맡기면 약 4 만개 정도의 transcripts read data가 나온다.
근데 어차피 거의 (내 경험상) illumina 장비로 한다.
그럼 이 결과물 중에 어떤 것에 초점을 맞추느냐는 목적에 따라 분석을 하면 된다.
나 같은 경우는, 예를 들자면, 신경계에서 어떤 gene을 upregulation 시키거나, 아니면 어떤 형식으로든 treatment를 하였을 때,
어떤 transcription factor가 움직이느냐를 주로 본다.
만약, 예를 들어 result에서 A 라는 trasncription factor의 RNA read 수가 높게 나왔다고 하면, 이것은 어떤 처치에 의해서 이 A의 activity가 많아졌다는 것이다.
당연한 이야기겠지만, transcripted RNA level 자체가 높아졌다는 것만으로도 의미는 있으며 추가적으로, ChIP과 같은 assay도 연계하여 살펴볼 수 있다.
간단하게, RNA-seq은 PCR을 하나하나 다 해보는 것이라 할 수 있다.
근데 이제 targeted primer로 하는 것이 아닌, 그냥 genome 전체를 sequencing해서 align하여 annotation을 붙인 것뿐이다.
Align하고 annotation을 한다는 개념이 Seq tech와 연관되어서 나오는 단어인데,
현재 범용적으로 쓰이는 sequencing 기술이 잘게 잘게 나뉘어진 gene들을 쭉 증폭시켜서 그것들을 하나로 이어붙인 다음 (align), 알려진 sequence에 맞추어서 annotation을 하는 것이기 때문이다
(https://www.youtube.com/watch?v=womKfikWlxM&ab_channel=Illumina).
예를 들어, RNA-seq 결과로 어떤 특정 gene에 대한 결과가 아주 좋게 나왔다고 하면, PCR로 자체적으로 confirm을 할 것이다.
https://www.youtube.com/watch?v=womKfikWlxM&ab_channel=Illumina" target="_blank" rel="noopener" data-mce-href="http:// http:// https://www.youtube.com/watch?v=womKfikWlxM&ab_channel=Illumina">http:// http:// https://www.youtube.com/watch?v=womKfikWlxM&ab_channel=Illumina
전체적인 RNA-seq 및 분석 flow는:
1) RNA-seq을 하려면 먼저, RNA를 준비해야 한다.
2) RNA sample을 준비하여 QC를 자체적으로 하고, sequencing 회사나 각 소속의 bioinformatics core lab에 의뢰한다.
3) Waiting 끝에 결과물을 받는다
4) R이나 Galaxy site 등을 사용하여 분석한다
5) 유의한 결과물을 도출하고 Figure를 만든다
즉, (내가 생각하기에) 결국 user 입장에서는
RNA sample 준비와 분석만 하면 된다.
거의 대다수의 경우, 기계나 장비로 직접 RNA-seq을 run할 일은 없을 것이다.
마치, 요즘 자체적으로 vector를 디자인은 하지만 생산은 company에 맡기듯이 😅
사실 분석도 illumina나 core lab에서 해주지만 ㅋㅋㅋ '내가 원하는' figure를 만들어준다던가 plot을 만들어 준다던가 까지는 해주지 않는다.
그건 이제 자신이 해야 한다.
기본적으로, illumina나 core lab이나 Gene Id를 change해주고 read count를 TPM or RPKM change까지는 해줬던 것 같다.
근데 왜 core lab 이야기를 계속 적냐면, 미국의 경우 자체 학교 core lab에 맡기는 게 결과가 경험상 더 빨리 나온다.
그리고 나름 건너건너 친분이 어느 정도는 있어서 relatively fast communication이 가능하다 (result analysis에 대한 discussion이 가능).
Illumina 같은 회사야 돈을 더 내면 더 빨리, 더 high qual로 나오긴 하지만, 대부분 default price로 default option인 분석을 그것도 할인까지 해서 맡기는 경우가 default일 것이다 (왜냐면 비쌈).
그리고 왜 illumina라는 회사의 이름이 계속 언급되냐면, 현재 sequencing paradigm을 가지고 있는 tech를 만든 회사이고 거의 독점적 위치를 가지고 있기 때문이다.
RNA sample을 준비하는 과정이나 protocol은 Nature protocol 등에서 매우 매우 자세하게 Note까지 붙여가면서 설명을 해놓았으니 아래 예시와 같은 paper를 참고하여 준비하면 된다.
더욱이, Tagging된 RNA만을 분석하는 게 더 유의미한 분석이 되므로 tagging 방법도 잘 알아보고 하는 것을 추천한다.
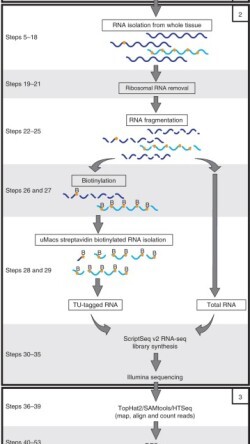
https://www.nature.com/articles/nprot.2014.085
Sampling이 끝나면 sequencing을 보내기 전에 QC는 self로 해주어야 하는데 RIN score라는 것이 있다.
1~10 score로 자동적으로 분석해 주는데 보통 Agilent의 kit로 분석해 주면 된다.
10점에 가까울수록 좋은 quality이고, 보통 8 점 이상의 sample을 보내게 되어 있다.
https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-rna-kits-reagents/bioanalyzer-rna-analysis-228256" target="_blank" rel="noopener" data-mce-href="http:// https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-rna-kits-reagents/bioanalyzer-rna-analysis-228256">http:// https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-rna-kits-reagents/bioanalyzer-rna-analysis-228256
RNA Electrophoresis, RNA Integrity, Bioanalyzer RIN | 애질런트
다른 고객들도 다음 제품들을 검색하셨습니다
www.agilent.com
Sample의 QC도 끝나면 sample을 보낼 곳에 deliver 해주고 기다리면,
seq 회사나 core lab에서 result data를 보내준다.
Raw data가 있고 summary result가 있는데 여하튼 합치면 굉장한 용량이라서 보통 기간제 cloud link를 보내준다.
그럼 다운 받을 것을 다운 받고 이제 입맛에 맞게 분석을 하면 되는데,
문제는 여기서부터는 이야기가 길어진다.
제공받을 summary file에는
[ID] [log2FoldChange] [pvalue] [padj] [sample reads] [Gene.name]
요렇게 적혀서 온다.
이걸로도 괜찮으면 가공해서 사용하면 되지만,
다른 방법으로도 하고 싶다 하면 이제 raw dat BAM file을 가지고 분석을 self로 잘 해봐야 한다.
여기서 프로그램을 좀 쓴다 하는 사람들은 R이나 기타 프로그램으로 자체적으로 분석을 하면 되는데 이런 사람들의 경우는 이미 숙련자들이라 해당사항이 없고,
나 같이 간간히 쓸 light user는 galaxy라는 site에서 해결을 하는 경우가 많을 것이다 (추측).
해당 site에 data를 올리고 각각의 step을 진행하면 align 및 DEseq까지 진행할 수 있다.
Cloud system이라 내 컴퓨터 공간을 차지하는 것도 아니고 클릭만으로 전부 다 가능하게 되어 있다.
Step에 대한 guide도 Galaxy에서 아주~ 친절하게 잘 설명해놓았다.
DEseq까지 진행을 했다 치면 이제 plot을 만들면 일단락의 분석은 했다고 볼 수 있다.
Plot은 보통 일반적으로 heatmap과 volcano plot을 base로 자주 볼 수 있을 것이다.
그런데 결국 heatmap과 volcano plot을 만들려면 R과 같은 프로그램으로 만들긴 해야 한다 😂😂😂😂
그래서 여차저차하여
result로서 유망한 gene을 추려낼 수 있을 것이다.
Upregulated gene A, B, C
Downregulated gene X, Y, Z
혹은 category에서 관련성 있는 gene들 중에서 a, b, c 등등
혹은 P value xxxx 이하의 gene list의 GO analysis를 통한 pick up gene x, y, z 등등
그럼 이제 이러한 정보를 바탕으로 다음 step의 실험을 진행할 수 있게 될 것이다.
'신경과학' 카테고리의 다른 글
| OPC의 synapse engulfment 에 대한 논문을 읽고 감상. 221007 (0) | 2022.10.07 |
|---|---|
| 우파루파 (axolotl) 신경재생 관련 snRNA-seq 논문을 읽고. 220909 (0) | 2022.09.13 |
| [번역] Neural correlates of consciousness (7/完) (0) | 2022.07.01 |
| [번역] Neural correlates of consciousness (6) (0) | 2022.06.26 |
| [번역] Neural correlates of consciousness (5) (0) | 2022.06.25 |